Características del VIH
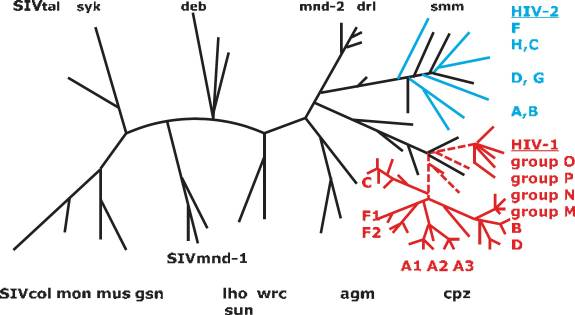
El virus de la inmunodeficiencia humana se agrupa en el género Lentivirus dentro de la familia Retroviridae, subfamilia Orthoretrovirinae. Según las características genéticas y las diferencias en los antígenos virales, el VIH se clasifica en los tipos 1 y 2 (VIH-1, VIH-2). Los virus de inmunodeficiencia de primates no humanos (virus de inmunodeficiencia de simios, VIS) también se agrupan en el género Lentivirus. Los análisis epidemiológicos y filogenéticos actualmente disponibles implican que el VIH se introdujo en la población humana entre 1920 y 1940. El VIH-1 evolucionó a partir de virus de inmunodeficiencia de primates no humanos de los chimpancés de África Central y el VIH-2 de los mangabeys hollín de África Occidental.
Estructura del genoma
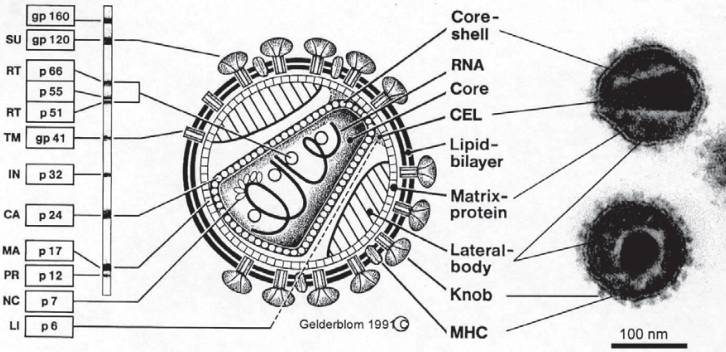
El genoma del VIH consta de dos moléculas idénticas de ARN monocatenario que están encerradas dentro del núcleo de la partícula viral. El genoma del provirus del VIH, también conocido como ADN proviral, se genera mediante la transcripción inversa del genoma del ARN viral en ADN, la degradación del ARN y la integración del ADN bicatenario del VIH en el genoma humano. El genoma del ADN está flanqueado en ambos extremos por secuencias LTR (repetición terminal larga). La región 5′ LTR codifica el promotor de la transcripción de los genes virales. En dirección 5 ‘ a 3’ sigue el marco de lectura del gen gag, que codifica las proteínas de la membrana central externa (MA, p17), la proteína de la cápsida (CA, p24), la nucleocápside (NC, p7) y una proteína más pequeña, proteína estabilizadora de ácidos nucleicos.
Al marco de lectura gag le sigue el marco de lectura pol que codifica las enzimas proteasa (PR, p12), transcriptasa inversa (RT, p51) y RNasa H (p15) o RT más RNasa H e integrasa (IN, p32). Adyacente al gen pol, sigue el marco de lectura env del que se derivan las dos glicoproteínas de la envoltura gp120 (proteína de superficie, SU) y gp41 (proteína transmembrana, TM).
Además de las proteínas estructurales, el genoma del VIH codifica varias proteínas reguladoras: Tat (proteína transactivadora) y Rev (regulador de empalme de ARN) son necesarias para el inicio de la replicación del VIH, mientras que las otras proteínas reguladoras Nef (factor regulador negativo), Vif (factor de infectividad viral), Vpr (proteína viral r) y Vpu (proteína viral única) tienen un impacto en la replicación viral, la gemación viral y la patogénesis. El VIH-2 codifica Vpx (proteína del virus x) en lugar de Vpu, que es parcialmente responsable de la reducida patogenicidad del VIH-2. La estructura del genoma de los virus de inmunodeficiencia de chimpancés y gorilas es idéntica a la del VIH-1.
Grupos y subtipos
El VIH-1 se subdivide en los grupos M, N, O y P. Sobre la base del análisis filogenético, se pueden asignar varios virus de chimpancé entre los grupos N y O de humanos y el SIV de gorilas en el grupo P. No está resuelto si el virus del chimpancé del grupo P se transmitió al ser humano directamente desde el chimpancé o desde el gorila.
Los virus VIH-1 del grupo M se subdividen en los subtipos A a D, F a H, J y K. En términos evolutivos, los grupos A y D parecen ser los virus más antiguos. Los subtipos B y D están muy relacionados y, por lo tanto, se consideran subtipos.
La recombinación, es decir, el intercambio de secuencias genéticas enteras en posiciones no seleccionadas se observa cuando una célula diana está infectada con dos subtipos diferentes de VIH. Estadísticamente, aproximadamente 1 de cada 400 partículas de virus recién producidas es recombinante. Se desarrollan cepas de virus genéticamente nuevas mediante recombinación que pueden tener una ventaja de selección dentro del huésped.
Estructura de partículas
La partícula madura del VIH es redonda, mide aproximadamente 100 nm de diámetro y tiene una membrana lipídica externa como envoltura. La envoltura contiene 72 protuberancias, compuestas por trímeros de las proteínas Env. Los trímeros de la proteína de superficie (SU) gp120 están anclados a la membrana mediante los trímeros de la proteína transmembrana gp41. Los epítopos neutralizantes dependientes de la conformación se encuentran en la proteína gp120. Estos están presentes en la proteína nativa, pero sólo se expresan parcialmente en la proteína desnaturalizada desplegada. La envoltura viral está compuesta por una bicapa lipídica y, en las partículas virales maduras, las proteínas de la envoltura SU y TM. Cubre la membrana exterior simétrica de la cápside formada por la proteína de la matriz (MA, p17). La cápside cónica se ensambla a partir de la proteína p24 (CA) de la cápside interna. Dependiendo del plano de sección, la cápside aparece como un cono, un anillo o una elipse en las micrografías electrónicas. El polo cónico de la cápside está unido a la membrana exterior de la cápside. Dos moléculas idénticas de ARN genómico viral se encuentran dentro de la cápside y varias moléculas de las enzimas virales RT/RNasa H e IN unidas al ácido nucleico. También están presentes en las partículas virales oligopéptidos que se generan después de la liberación de la célula durante la maduración de los viriones mediante el procesamiento proteolítico de las proteínas precursoras.
La maduración de la partícula de VIH tiene lugar mediante la escisión de las proteínas precursoras Gag y Gag/Pol (p55, p160) en proteínas individuales al final del proceso de gemación y durante la liberación de los viriones de la célula.
Infección de células humanas
Los pasos iniciales de la infección de una célula se caracterizan por complejas interacciones proteína-proteína. La glicoproteína de superficie gp120 de la partícula madura del VIH se une al receptor CD4 de la célula huésped. Todas las células CD4 positivas, como las células T colaboradoras, los macrófagos, las células dendríticas y los astrocitos, son susceptibles al VIH. Después de la unión a la molécula CD4 a través del dominio C4 de gp120, se produce un cambio conformacional en CD4 y gp120, lo que abre un sitio adicional para que gp120 permita la unión al correceptor, es decir, al receptor de quimiocina 5 (CCR5) o al receptor de quimiocina 4. (CXCR4 o fusina) en la superficie celular. La unión de gp120 a CD4 y al correceptor desencadena un cambio conformacional adicional en gp120 y posteriormente en gp41. El extremo N de gp41 se presenta en la membrana viral, forma un canal y, debido a su alta hidrofobicidad, se inserta en la membrana plasmática de la célula diana. Luego se completa la fusión de la membrana celular y la envoltura viral.
La fusión de las membranas viral y celular conduce a la translocación de la cápside viral al citoplasma. La cápside es absorbida por un endosoma y un cambio en el valor del pH en el fagosoma induce la liberación del contenido de la cápside en el citoplasma. La activación de la transcriptasa inversa (RT) tiene lugar en el citoplasma. La RT del VIH transcribe el genoma del ARN del VIH monocatenario en ADN (ADN complementario o ADNc). Paralelamente a la síntesis de ADN, la cadena de ARN se degrada enzimáticamente por la RNasa H, seguida de la conversión de ADNc monocatenario en ADN bicatenario (ADN proviral) mediante la actividad ADN polimerasa dependiente de ADN de RT. Este ADN proviral se transporta a través de nucleoporos al núcleo celular en forma de un complejo formado por la integrasa (IN) y el ADN proviral lineal o circular. Luego, la integrasa inserta al azar el genoma proviral en el genoma de la célula huésped humana. La integración del ADN proviral finaliza la infección por VIH de la célula y el establecimiento de una infección persistente.
El genoma proviral puede replicarse junto con el genoma de la célula huésped y como parte de él durante la división celular (infección latente que parece ser rara). Sin embargo, después de la activación de las células infectadas, el promotor LTR del genoma proviral puede servir como sitio de unión para las ARN polimerasas celulares dependientes de ADN y una variedad de factores de transcripción que inician la síntesis de ARNm viral y ARN genómico. La síntesis de ARNm viral de longitud completa está regulada, entre otros, por Tat y acelerada al máximo por Tat (p14).
La unión del VIH a una célula CD4 positiva requiere alrededor de 30 min a 2 h, la transcripción del genoma de ARN viral en ADN proviral se completa después de aproximadamente 6 h y la integración en el genoma del huésped tarda 6 h adicionales. Después de la integración, las primeras partículas de virus son detectables después de aproximadamente 12 h; es decir, aproximadamente 24 h después de la infección, los primeros virus descendientes se liberan de la célula infectada. Debido a que la RT viral no tiene actividad correctora, estadísticamente se incorpora al ADN proviral un nucleótido incorrecto por ronda de transcripción. Si la replicación del VIH no está restringida, se espera un recambio diario de 10 8 -10 9 partículas virales, es decir, recién producidas por células infectadas y destruidas por el sistema inmunológico. Suponiendo una tasa de mutación de 1 en 10 4 nucleótidos por genoma durante un ciclo de replicación, con el tiempo puede desarrollarse en un paciente un amplio espectro de diversas cuasiespecies. Dado que los epítopos de los anticuerpos neutralizantes también se ven afectados por la mutación, estas cuasiespecies pueden evadir continuamente el sistema inmunológico, infectar nuevas células y, por lo tanto, mantener la producción de VIH. No todos los cambios de nucleótidos resultan en el intercambio de un aminoácido. Sin embargo, las mutaciones en regiones esenciales de las proteínas estructurales o en los centros activos de las enzimas que influyen dan lugar a mutantes del VIH incompetentes para la replicación.
Los linfocitos T infectados se eliminan con una vida media de 2 a 4 días de la sangre de una persona infectada por el VIH mediante componentes citotóxicos del VIH, lisis de células productoras de virus o mediante linfocitos T citotóxicos como parte de la respuesta inmune. Dado que las células T colaboradoras infectadas por el VIH también se lisan y al mismo tiempo se inhibe la producción de dichas células, se observa una disminución gradual de las células T colaboradoras. Las proteínas específicas del VIH como Nef y Tat son responsables de la maduración y el reemplazo insuficientes de las células T auxiliares. Por lo tanto, parte de los linfocitos T auxiliares recién producidos no desarrollan funciones normales. Después de una infección por VIH de larga duración, la pérdida continua de linfocitos T cooperadores provoca una inmunodeficiencia. El VIH integrado en el genoma del huésped de células de larga vida como macrófagos, astrocitos o células T de memoria puede persistir en fase latente durante varios años (la vida media de determinadas células diana es de 7 años). La activación de dichas células da como resultado la producción de partículas infecciosas de VIH.
Fuente
German Advisory Committee Blood (Arbeitskreis Blut), Subgroup ‘Assessment of Pathogens Transmissible by Blood’. Human Immunodeficiency Virus (HIV). Transfus Med Hemother. 2016 May;43(3):203-22. doi: 10.1159/000445852. Epub 2016 May 9. PMID: 27403093; PMCID: PMC4924471.